Varidnaviria -Varidnaviria
| Varidnaviria | |
|---|---|

|
|
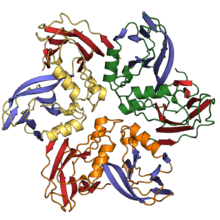
| Un diagramma a nastro del DJR-MCP del virus Pseudoalteromonas PM2 , con le due pieghe del rotolo di gelatina colorate in rosso e blu | |
|
Classificazione dei virus |
|
| (non classificato): | Virus |
| regno : | Varidnaviria |
| Subtaxa | |
| Sinonimi | |
|
|
Varidnaviria è un regno di virus che include tutti i virus a DNA che codificano le principali proteine del capside che contengono una piega verticale di gelatina . Le principali proteine del capside (MCP) si formano in subunità pseudoesamerica del capside virale, che immagazzina l' acido desossiribonucleico virale(DNA), e sono perpendicolari, o verticali, alla superficie del capside. Oltre a questo, i virus nel regno condividono anche molte altre caratteristiche, come le proteine minori del capside (mCP) con la piega verticale del rotolo di gelatina, un'ATPasi cheimpacchetta ilDNA virale nel capside e una DNA polimerasi che replica il genoma virale.
Varidnaviria è stata fondata nel 2019 sulla base delle caratteristiche condivise dei virus nel regno. Ci sono due gruppi di virus in Varidnaviria : virus che hanno una doppia piega verticale gelatina (DJR) nell'MCP, assegnati al regno Bamfordvirae , e virus che hanno una singola piega verticale gelatina (SJR) nell'MCP , assegnati a il regno degli Elvetiaviri . Si pensa che il lignaggio DJR-MCP discenda dal lignaggio SJR-MCP tramite un evento di fusione genica e l'SJR-MCP mostra una stretta relazione con le nucleoplasmine , indicando una possibile origine dell'MCP della piega del rotolo di gelatina del regno. La maggior parte dei virus a DNA eucariotici identificati appartengono a Varidnaviria .
I virus marini nel regno sono molto abbondanti in tutto il mondo e sono importanti nell'ecologia marina. Molti virus animali nel regno sono associati a malattie, inclusi adenovirus , poxvirus e il virus della peste suina africana . I poxvirus sono stati importanti nella storia della medicina, in particolare il vaiolo , causato dal virus Variola , che fu il bersaglio del primo vaccino e che in seguito divenne la prima malattia debellata. Il regno include anche virus giganti che sono fisicamente più grandi e contengono un numero molto maggiore di geni rispetto ai virus tipici.
Etimologia
Il nome " Varidnaviria " è un portmanteau di Vari unità organizzative del DNA virus e il suffisso - viria , che è il suffisso usato per regni virus. I virus a DNA a doppio filamento (dsDNA) nel regno sono spesso chiamati virus dsDNA senza coda o senza coda per distinguerli dai virus dsDNA a coda di Duplodnaviria .
Caratteristiche
MCP, mCP e ATPasi
La maggior parte dei virus in Varidnaviria contiene un capside costituito dalle principali proteine del capside che contengono pieghe verticali singole (SJR) o doppie gelatine (DJR). Le principali proteine del capside sono chiamate così perché sono le proteine primarie di cui è fatto il capside. Una piega del rotolo di gelatina è un tipo di struttura piegata in una proteina in cui otto filamenti beta antiparalleli sono organizzati in quattro fogli beta antiparalleli in un layout simile a un rotolo di gelatina , chiamato anche rotolo svizzero. Ciascun filamento beta è una sequenza specifica di amminoacidi e questi filamenti si legano ai loro filamenti antiparalleli tramite legami idrogeno . La differenza tra le pieghe SJR e DJR è che una piega DJR è semplicemente due pieghe SJR in una singola proteina. Le pieghe verticali sono quelle perpendicolari alla superficie del capside, in contrasto con le pieghe orizzontali parallele alla superficie del capside.
Durante il processo di assemblaggio del capside virale, gli MCP si autoassemblano in strutture esagonali, chiamate esoni, contenenti più copie dell'MCP. Gli esoni si legano quindi per formare i lati triangolari relativamente piatti del capside icosaedrico. Tutti i virus in Varidnaviria che codificano un DJR-MCP che sono stati analizzati in alta risoluzione codificano anche una proteina del capside minore (mCP) che contiene una piega SJR. Questi mCP si assemblano in strutture pentagonali chiamate pentoni che formano i vertici pentagonali del capside icosaedrico.
La maggior parte dei membri del regno codifica anche ATPasi di confezionamento del genoma della superfamiglia FtsK-HerA. Le ATPasi in Varidnaviria sono enzimi che impacchettano il DNA virale nel capside durante il processo di assemblaggio dei virioni. FtsK è una famiglia di proteine che contiene una proteina transmembrana con quattro eliche che attraversano la membrana all'inizio della sequenza amminoacidica della proteina e un'ATPasi con una piega P-loop alla fine della sequenza amminoacidica della proteina , e la famiglia HerA è omologa a FtsK. L'esatta funzione dell'ATPasi per alcuni virus in Varidnaviria non è chiara poiché le caratteristiche morfologiche, come il genoma circolare e superavvolto del virus Pseudoalteromonas PM2 , sembrano proibire la traslocazione da parte dell'ATPasi del DNA dall'esterno del capside verso l'interno. Il sottoinsieme della superfamiglia FtsK-HerA trovato in Varidnaviria è spesso chiamato clade A32, dal nome del gene A32(R) codificante per l'ATPasi del virus Vaccinia .
Altre caratteristiche
A parte la triade morfogenetica di base dei geni, MCP, mCP e ATPasi, alcune altre caratteristiche sono comuni o uniche in vari lignaggi all'interno di Varidnaviria , elencati di seguito.
- Molti membri del regno codificano per una DNA polimerasi di tipo B, che copia il DNA virale e spesso componenti aggiuntivi della DNA polimerasi, come la superfamiglia 3 elicasi , o proteine di inizio della replicazione, nel caso della famiglia Corticoviridae . Un'eccezione è l'ordine Halopanivirales , i cui membri non codificano alcun enzima di replicazione riconoscibile.
- Molti virus eucarioti DJR-MCP codificano una proteasi di maturazione del capside che è coinvolta nell'assemblaggio del capside.
- Alcuni membri del regno codificano per l' integrasi , un enzima che integra il genoma virale nel genoma dell'ospite.
- La maggior parte dei membri del regno ha capsidi a forma di icosaedro, contenente 20 facce triangolari e 12 vertici.
- In vari lignaggi, inclusi ascovirus e poxvirus, la forma icosaedrica ancestrale del capside è stata persa e sostituita con altre forme, come ovoidali e simili a mattoni.
- I poxvirus codificano una proteina scaffold , che guida la costruzione geometrica del capside virale, che si ripiega anche negli pseudo-esameri DJR.
- Alcuni virus hanno vertici speciali nei loro capsidi icosaedrici per trasportare il genoma fuori dal capside e per creare fabbriche di virus .
- Per alcuni virus, il genoma all'interno del capside è circondato da una membrana lipidica .
- Quasi tutti i virus DJR-MCP riconosciuti codificano un ATPase della superfamiglia FtsK-HerA. Gli adenovirus sono l'eccezione, codificando invece la propria ATPasi distinta che ha lo stesso ruolo dell'ATPasi FtsK-HerA.
- La famiglia Finnlakeviridae e un gruppo provvisorio chiamato gruppo Odin, entrambi membri proposti di Varidnaviria , mancano della superfamiglia ATPase FtsK-HerA.
- Tutti i membri di Varidnaviria ad eccezione di Finnlakeviridae , una famiglia membro proposta, hanno genomi dsDNA. I virus in Finnlakeviridae hanno invece genomi di DNA a singolo filamento (ssDNA).
filogenetica
È stato suggerito che Varidnaviria sia precedente all'ultimo antenato comune universale (LUCA) della vita cellulare e che i virus del regno fossero presenti nel LUCA. Le SJR-MCP verticali di Halopanivirales , assegnate al regno Helvetiavirae , a differenza delle pieghe SJR trovate al di fuori della Varidnaviria , mostrano una relazione con un gruppo di proteine che include la superfamiglia Cupin e le nucleoplasmine, indicando una possibile origine della principale proteina del capside della Varidnaviria tra questo gruppo. Il lignaggio DJR-MCP, assegnato al regno Bamfordvirae , da allora in poi sembra essere venuto all'esistenza per mezzo di un evento di fusione genica che ha unito i due SJR-MCP in uno, indicato dai due SJR-MCP che formano un reticolo nel capside che assomiglia strutturalmente al reticolo del capside DJR-MCP. I virus dsDNA archeali in Portogloboviridae contengono solo un SJR-MCP verticale, che sembra essere stato duplicato in due per Halopanivirales , quindi l'MCP di Portogloboviridae rappresenta probabilmente uno stadio precedente nella storia evolutiva degli MCP di Varidnaviria .
I virus di Bamfordvirae sembrano essersi incrociati dai procarioti agli eucarioti all'inizio della storia eucariotica, tramite l'infezione da un tectivirus o da un virus simile al tectivirus di un batterio che è diventato un simbionte batterico in un proto-eucariota. Da lì, sulla base dell'analisi filogenetica della DNA polimerasi virale e di altre caratteristiche, i virus eucariotici in Bamfordvirae sembrano aver formato una complessa relazione con vari elementi genetici egoistici , inclusi i polinton , un tipo di trasposone , porzioni di DNA che possono auto-replicarsi e si integrano in altre parti della stessa molecola di DNA, e alcuni tipi di plasmidi , che sono molecole di DNA extra-cromosomiche che si autoreplicano all'interno della cellula o organello che occupano.
È probabile che il simbionte batterico iniziale sia diventato mitocondri, con i plasmidi lineari mitocondriali che discendono dai tectivirus rimanenti. Un altro lignaggio divergente ha raggiunto il nucleo e si è ricombinato con i trasposoni, diventando polintoni, che potrebbero essere stati i primi virus eucarioti in Bamfordvirae o correlati ai primi. I polinton hanno quindi dato origine a molteplici lignaggi mediante vari meccanismi. Tra questi lignaggi ci sono virus a tutti gli effetti, inclusi adenovirus e virus giganti, plasmidi lineari citoplasmatici , virofagi , che sono virus satelliti di virus giganti, transpovirons , che sono molecole di DNA simili a plasmidi lineari presenti nei virus giganti e bidnavirus tramite ricombinazione genetica con un parvovirus , entrambi classificati nel regno Monodnaviria .
Mentre la piega del rotolo di gelatina si trova in altri regni, inclusa la famiglia Microviridae in Monodnaviria e vari virus a RNA a filamento singolo in Riboviria , la piega del rotolo di gelatina trovata in Varidnaviria è verticale, cioè perpendicolare alla superficie del capside, contrariamente alle pieghe del rotolo di gelatina in altri regni, che sono orizzontali, cioè paralleli alla superficie del capside. In generale, gli altri regni dei virus non hanno una relazione apparente basata sulla comune discendenza alla Varidnaviria .
Classificazione
Varidnaviria ha due regni: Bamfordvirae e Helvetiavirae , l'ultimo dei quali è monotipico fino al rango di famiglia. Questa tassonomia può essere visualizzata come segue:
- Regno: Bamfordvirae , che codifica per una delle principali proteine del capside che contiene una piega verticale a doppio rotolo di gelatina
- Regno: Helvetiavirae , che codifica per una delle principali proteine del capside che contiene una singola piega di gelatina verticale
- Phylum: Dividoviricota
- Classe: Laserviriceti
- Ordine: Halopanivirales
- Classe: Laserviriceti
- Phylum: Dividoviricota
Tutti i membri riconosciuti di Varidnaviria appartengono al Gruppo I: virus dsDNA del sistema di classificazione di Baltimora , che raggruppa i virus in base a come producono l'RNA messaggero. La famiglia Finnlakeviridae , una proposta famiglia di Varidnaviria , appartiene al gruppo II: virus ssDNA e sarebbe l'unico virus ssDNA nel regno. La maggior parte dei virus a DNA identificati che infettano gli eucarioti appartengono a Varidnaviria , gli altri principali lignaggi di virus a DNA eucariotici sono l'ordine Herpesvirales , che infetta gli animali, in Duplodnaviria , e la classe Papovaviricetes , che infetta gli animali, in Monodnaviria . I regni sono il livello più alto di tassonomia utilizzata per i virus e Varidnaviria è uno dei quattro, gli altri tre sono Duplodnaviria , Monodnaviria e Riboviria .
La famiglia non assegnata Portogloboviridae è una famiglia proposta del regno poiché le sue proteine del capside sembrano essere omologhe a quelle dei virus in Varidnaviria .
Un altro gruppo proposto è la classe Naldaviricetes (incluso Polydnaviridae ). Questi virus comprendono diversi geni che sono lontanamente correlati ai geni centrali del Nucleocytoviricota e quindi potrebbero essere membri altamente derivati dei virus DJR-MCP, nonostante l'assenza del DJR-MCP e la formazione di virioni di forma strana. L'analisi filogenetica preliminare di diversi geni essenziali condivisi da tutti questi virus artropodi e dai Nucleocytoviricota , come PolB, subunità RNAP, elicasi-primasi e tiolo ossidoreduttasi, ha suggerito che questo gruppo di virus potrebbe essere una derivazione altamente derivata del Nucleocytoviricota .
Interazioni con gli host
Malattia
I batteriofagi in Varidnaviria sono potenzialmente una delle principali cause di morte tra i procarioti marini . Questo punto di vista si basa su autolicivirus che hanno un'ampia gamma di ospiti, che infettano e uccidono molti ceppi diversi di varie specie di batteri, in contrasto con i batteriofagi muniti di coda, che hanno intervalli di ospiti più limitati, nonché sul numero apparentemente elevato di virus dsDNA marini senza coda. . I virus algali della famiglia Phycodnaviridae svolgono un ruolo importante nel controllo della proliferazione algale e, con molti virus marini in generale, contribuiscono a un processo chiamato shunt virale , per cui il materiale organico proveniente da organismi uccisi viene "sviato" dai virus lontano dai livelli trofici più elevati e riciclati per il consumo da quelli a livelli trofici inferiori.
I virus che causano malattie più importanti nella Varidnaviria sono gli adenovirus, i poxvirus e il virus della peste suina africana (ASFV). Gli adenovirus in genere causano lievi malattie respiratorie, gastrointestinali e congiuntivali, ma occasionalmente causano malattie più gravi, come la cistite emorragica , l' epatite e la meningoencefalite . I poxvirus infettano molti animali e in genere causano sintomi non specifici associati a una caratteristica eruzione cutanea chiamata vaiolo. Poxvirus notevoli includono il virus Variola , che causa il vaiolo e il virus Vaccinia , che viene utilizzato come vaccino contro il vaiolo. La PSA è solitamente asintomatica nei suoi serbatoi naturali, ma provoca una febbre emorragica letale nei suini domestici che è una preoccupazione per la produzione agricola.
endogenizzazione
Molti virus in Varidnaviria codificano per l'enzima integrasi, consentendo loro di integrare il loro genoma nel loro ospite e di comportarsi come trasposoni. I polinton strettamente correlati sono apparentemente sempre endogeni nei loro ospiti. Questa integrazione del DNA virale nel genoma dell'ospite è una forma di trasferimento genico orizzontale tra organismi non correlati, sebbene i polintoni siano tipicamente trasmessi verticalmente da genitore a figlio.
Immunità adattativa
Un esempio peculiare di endogenizzazione in Varidnaviria sono i virofagi, virus satelliti che dipendono dall'infezione da virus gigante per replicarsi. I virofagi si replicano dirottando gli apparati di replicazione di virus giganti, sopprimendo così il numero di virioni di virus giganti prodotti, aumentando la probabilità di sopravvivenza dell'ospite. Alcuni virofagi sono in grado di diventare endogeni e questa endogenizzazione può essere considerata una forma di immunità adattativa per l'ospite contro l'infezione da virus gigante.
Storia
Le malattie causate dai poxvirus sono note da gran parte della storia documentata. Il vaiolo, in particolare, è stato molto importante nella medicina moderna; il primo vaccino ad essere inventato mirava al vaiolo, e il vaiolo sarebbe poi diventato la prima malattia ad essere debellata. Gli adenovirus umani sono stati i primi virus DJR-MCP in Varidnaviria ad aver analizzato i loro MCP, distinguendosi per avere pieghe del rotolo di gelatina perpendicolari, piuttosto che parallele, alla superficie del capside. Nel 1999, la struttura dell'MCP del virus Pseudomonas PRD1 è stata risolta, dimostrando che il lignaggio DJR-MCP includeva virus procarioti. Il virus Haloarcula hispanica SH1 sarebbe poi diventato, nel 2003, il primo virus SJR-MCP scoperto.
Nel tempo, l'uso della metagenomica ha consentito l'identificazione di virus nell'ambiente anche senza l'identificazione dell'ospite o di campioni di laboratorio, portando alla scoperta di molti membri aggiuntivi del regno. Indagini morfologiche di campioni marini suggeriscono che i virus dsDNA senza coda potrebbero essere più numerosi dei virus dsDNA con coda di Duplodnaviria , che, a partire dal 2019, sono la linea di virus più ampia e diversificata documentata. Con la maggiore conoscenza dei virus del regno, Varidnaviria è stata fondata nel 2019 sulla base dei tratti condivisi dei virus nel regno.
L'istituzione di Varidnaviria ha consentito di classificare virus recentemente scoperti e correlati, ma divergenti, in taxa più elevati. Ciò include le famiglie proposte come Finnlakeviridae , che sarebbe l'unica famiglia nel regno con un genoma di DNA a filamento singolo, Autolykiviridae , che hanno una vasta gamma di ospiti e che possono svolgere un ruolo importante nella morte dei batteri marini , e il " Odin", che codifica per una proteina che non ha alcuna relazione nota con altre proteine al posto della superfamiglia ATPasi FtsK-HerA. Nel 2020, gli autolicivirus sono stati ufficialmente classificati per la prima volta.
Guarda anche
Appunti
Riferimenti
Ulteriori letture
- Ward, CW (1993). "Progressi verso una più alta tassonomia dei virus" . Ricerca in virologia . 144 (6): 419–53. doi : 10.1016/S0923-2516(06)80059-2 . PMC 7135741 . PMID 8140287 .